
蛋白质结构的解析对于生物科学的多个领域至关重要,包括药物设计、生物技术和疾病研究。然而,随着蛋白质结构数据的迅速积累,如何高效地分析这些数据成为一个挑战。近年来,新开发的Foldseek Cluster算法在这一领域取得了重大突破,使得大规模蛋白结构分析成为可能。
Foldseek Cluster算法的背景
Foldseek Cluster算法是一种基于机器学习和大数据技术的新型蛋白结构分析工具。它的开发旨在解决传统方法在处理大规模蛋白结构数据时效率低下的问题。该算法采用了多种数据挖掘和模式识别技术,能够自动识别、分类和分析蛋白质结构,从而加速了蛋白质结构研究的进程。
Foldseek Cluster算法的关键特性
1.高速分析:Foldseek Cluster算法能够一次性分析大量的蛋白结构数据,将传统的手工分析过程大幅缩短。
2.自动化:该算法实现了蛋白质结构分析的自动化,减少了研究人员的人力投入,同时提高了分析的一致性和准确性。
3.多样性数据处理:Foldseek Cluster不仅能够处理X射线晶体学数据,还可以应用于核磁共振(NMR)和电子显微镜等不同类型的蛋白质结构数据。
4.模式识别:该算法采用了先进的模式识别技术,有助于发现潜在的蛋白质结构模式和趋势,从而深化对蛋白质结构的理解。
科研应用和前景
Foldseek Cluster算法的应用前景广泛。它可以加速新蛋白质结构的发现,有助于揭示蛋白质结构与功能之间的关系,促进了药物设计、酶工程和疾病研究等领域的发展。此外,该算法还为科学家提供了更多的工具,用于解答蛋白质折叠、动力学和相互作用等重要问题。
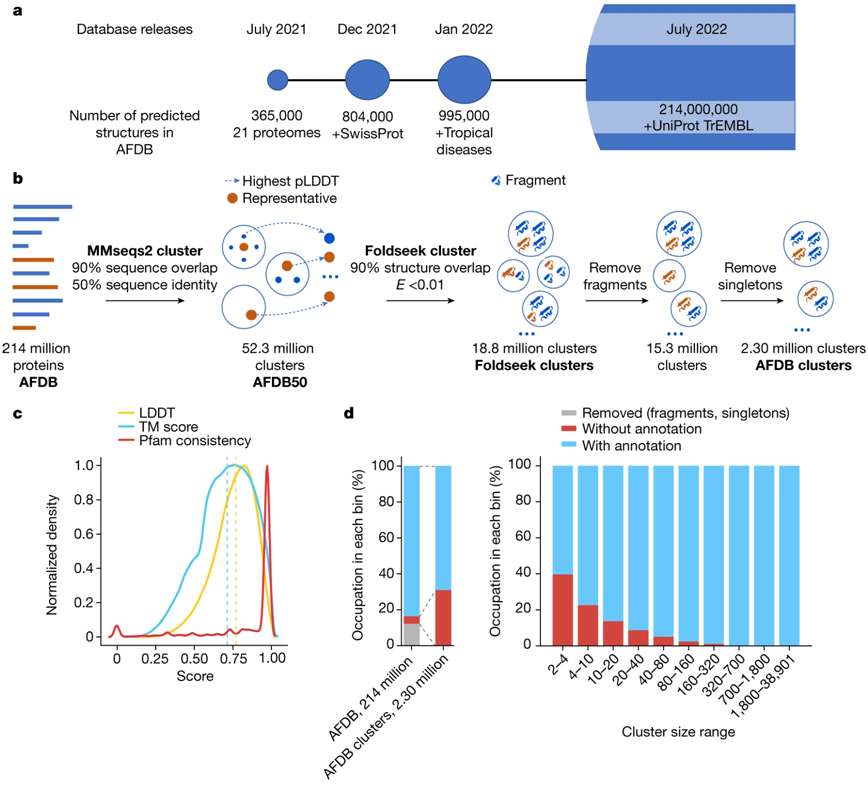
▲AlphaFold数据库,结构聚类工作流程和结构簇
结论
Foldseek Cluster算法的出现标志着蛋白结构分析领域的一次革命。它不仅加速了蛋白质结构数据的处理,还为科学家们提供了更多的研究可能性。这一算法的不断发展和应用将为生命科学领域带来更多的突破和创新,有望改善人类健康、生物技术和药物研发等方面的重要问题。 Foldseek Cluster算法的未来是令人兴奋的,将为蛋白质结构研究领域带来新的可能性。


